|
|
| (31 versions intermédiaires par 14 utilisateurs non affichées) |
| Ligne 3 : |
Ligne 3 : |
|
| |
|
| ''Les domaines sont rangés en ordre alphabétique'' | | ''Les domaines sont rangés en ordre alphabétique'' |
| | |
| | ===Logiciels libres pour le traitement d'images=== |
| | |
| | * [http://rsbweb.nih.gov/ij/ ImageJ] (domaine public) |
|
| |
|
| ===Logiciels libres pour la Biologie=== | | ===Logiciels libres pour la Biologie=== |
| Ligne 17 : |
Ligne 21 : |
| ====Logiciels et banques de données dédiés aux séquences==== | | ====Logiciels et banques de données dédiés aux séquences==== |
|
| |
|
| [http://wiki.april.org/w/Science/LLbio_seq Ici] sont listés les logiciels libres spécialement dédiés aux différents traitements de séquences nucléotidiques et protéiques : recherche par homologie, alignement local/global, de deux séquences/multiple,... | | [[Science/LLbio_seq|Ici]] sont listés les logiciels libres spécialement dédiés aux différents traitements de séquences nucléotidiques et protéiques : recherche par homologie, alignement local/global, de deux séquences/multiple,... |
|
| |
|
| ====Logiciels et banques de données dédiés aux structures==== | | ====Logiciels et banques de données dédiés aux structures==== |
|
| |
|
| [http://wiki.april.org/w/Science/LLbio_struct Ici] sont présents quelques liens vers des logiciels et banques gérant les structures moléculaires (protéines, ARN,...). | | [[Science/LLbio_struct|Ici]] sont présents quelques liens vers des logiciels et banques gérant les structures moléculaires (protéines, ARN,...). |
|
| |
|
| ====Logiciels dédiés à la reconstruction phylogénétique==== | | ====Logiciels dédiés à la reconstruction phylogénétique==== |
|
| |
|
| Les puristes remarqueront que je n'ai pas parlé d'arbre ;-) Donc, pour l'inférence d'histoires évolutives, c'est par [http://wiki.april.org/w/Science/LLbio_phylo ici]. | | Les puristes remarqueront que je n'ai pas parlé d'arbre ;-) Donc, pour l'inférence d'histoires évolutives, c'est par [[Science/LLbio_phylo|ici]]. |
|
| |
|
| ====Logiciels et banques de données pour les données d'expression==== | | ====Logiciels et banques de données pour les données d'expression==== |
|
| |
|
| Quelques logiciels et ressources publiques sont mentionnés [http://wiki.april.org/w/Science/LLbio_array ici] : il s'agit d'outils servant à analyser et contenir les données de puces à ADN (lames de verre, Affymetrix, etc.). | | Quelques logiciels et ressources publiques sont mentionnés [[Science/LLbio_array|ici]] : il s'agit d'outils servant à analyser et contenir les données de puces à ADN (lames de verre, Affymetrix, etc.). |
|
| |
|
| ====Logiciels pour la visualisation====
| | ===Logiciels pour la visualisation=== |
|
| |
|
| Parce qu'il faut bien faire de [http://wiki.april.org/w/Science/LLbio_visualisation jolies zimages] :-) Ce n'est pas la seule raison, loin s'en faut : donner un aperçu en images intelligible à sa recherche est très important. | | Parce qu'il faut bien faire de [[Science/LLbio_visualisation|jolies zimages]] :-) Ce n'est pas la seule raison, loin s'en faut : donner un aperçu en images intelligible à sa recherche est très important. |
| | |
| | * [http://www.gnuplot.info/ gnuplot] (non libre au sens de la FSF. Les modifications doivent être redistribuées sous forme de patch uniquement.) |
| | * [http://www.pyxplot.org.uk/ PyXPlot] |
| | * [http://matplotlib.sourceforge.net/ matplotlib] |
| | * [http://k3dsurf.sourceforge.net/index_fr.html K3DSurf] |
| | * [http://mayavi.sourceforge.net/ MayaVi] |
| | * [http://soft.proindependent.com/qtiplot.html QtiPlot] |
| | * [http://www.ggobi.org GGobi] Exploration de données à hautes dimensions |
| | * [http://www.slicer.org/ 3DSlicer] Visualisation et analyse d'imagerie médicale (license BSD-like) |
|
| |
|
| ===Logiciels libres pour la Chimie=== | | ===Logiciels libres pour la Chimie=== |
| Ligne 46 : |
Ligne 59 : |
| * [http://wxmaxima.sourceforge.net/wiki/index.php/Main_Page wxMaxima] | | * [http://wxmaxima.sourceforge.net/wiki/index.php/Main_Page wxMaxima] |
| * [http://www-fourier.ujf-grenoble.fr/~parisse/giac_fr.html Giac/Xcas] | | * [http://www-fourier.ujf-grenoble.fr/~parisse/giac_fr.html Giac/Xcas] |
| | * [http://cadabra.phi-sci.com cadabra] |
| | * Autres ''computer algebra systems'' (CAS) libres décris ici : http://www.cmla.ens-cachan.fr/fileadmin/Groupes/LaForge/Final/cas3.pdf |
|
| |
|
| ====Géométrie Algébrique==== | | ====Géométrie Algébrique==== |
| Ligne 57 : |
Ligne 72 : |
| ====Calcul Numérique==== | | ====Calcul Numérique==== |
|
| |
|
| [http://www.scilab.org scilab] | | * [http://www.scilab.org scilab] |
| | | * [http://www.gnu.org/software/octave/about.html octave] |
| [http://www.gnu.org/software/octave/about.html octave] | | * [http://www.sagemath.org/ sage] |
| | | * [http://numpy.scipy.org/ NumPy] |
| [http://www.sagemath.org/ sage] | | * [http://euler.sourceforge.net/ Euler] |
|
| |
|
| ==Logiciels libres pour la Physique== | | ==Logiciels libres pour la Physique== |
| ''(comprend aussi ceux pour l'Astronomie et la cosmologie)'' | | ''(comprend aussi ceux pour l'Astronomie et la cosmologie)'' |
| | |
| | * [http://www.shatters.net/celestia Celestia] |
| | * [http://www.stellarium.org/ Stellarium] |
|
| |
|
| ==Logiciels libres pour la Mécanique== | | ==Logiciels libres pour la Mécanique== |
| | |
| | ====Étude de vidéos de mouvements==== |
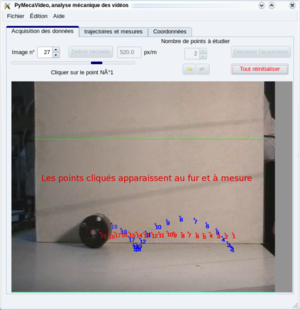
| | [http://outilsphysiques.tuxfamily.org/pmwiki.php/Oppl/Pymecavideo PyMecavideo] [[Image:pymecavideo2.png|thumb]] |
| | Ce que PyMecavideo peut faire : |
| | |
| | * charger une vidéo |
| | * pointer autant de points voulus (intéressant pour l'étude des référentiels et de la chute libre) |
| | * exporter la liste des point dans un format compréhensible (CSV) ou presse-papier |
| | * ... |
| | * peut jouer une vidéo du mouvement dans le référentiel choisi Exemple : si on pointe une balle et la roue du véolo depuis lequel la balle est lâchée, on peut voir le mouvement de la balle dans le référentiel du vélo. |
|
| |
|
| ====Eléments finis==== | | ====Eléments finis==== |
| Ligne 77 : |
Ligne 105 : |
|
| |
|
| [http://www.openfoam.com OpenFoam] | | [http://www.openfoam.com OpenFoam] |
| | |
| | [http://www.cfd-online.com/Wiki/Codes#Free_codes CFD-online] propose une liste plus complète de codes de CFD libres. |
|
| |
|
| ====Conduction et rayonnement thermique==== | | ====Conduction et rayonnement thermique==== |
| Ligne 82 : |
Ligne 112 : |
| [http://innovation.edf.com/recherche-et-communaute-scientifique/logiciels/syrthes-41220.html Syrthes] | | [http://innovation.edf.com/recherche-et-communaute-scientifique/logiciels/syrthes-41220.html Syrthes] |
|
| |
|
| ====Pré et post-traitement==== | | ====Pré- et post-traitement==== |
|
| |
|
| [http://www.salome-platform.org Salome] | | [http://www.salome-platform.org Salome] |
|
| |
|
| [http://www.paraview.org/ Paraview] | | [http://www.paraview.org/ Paraview] |
| | |
| | [http://www.geuz.org/gmsh/ GMSH] |
|
| |
|
| ====Distribution live==== | | ====Distribution live==== |
|
| |
|
| [http://www.caelinux.com CAElinux] | | [http://www.caelinux.com CAElinux]: distribution linux live permettant de tester simplement la plupart des logiciels cités ci-dessus. |
|
| |
|
| ==Logiciels libres pour la Psychologie et les neurosciences== | | ==Logiciels libres pour la Psychologie et les neurosciences== |
| Ligne 100 : |
Ligne 132 : |
| ''TODO : A vérifier où ça en est'' | | ''TODO : A vérifier où ça en est'' |
|
| |
|
| =Journaux libres pour la science= | | == Calcul distribué == |
| '''Partie en cours de modification'''
| |
| | |
| Publication ouverte en science
| |
| ''(à compléter)''
| |
| Source : [http://www.earlham.edu/~peters/fos/bethesda.htm Bethesda Statement on Open Access Publishing]
| |
|
| |
|
| ===PLoS===
| | [http://boinc.berkeley.edu/ BOINC] |
| [http://www.plos.org/index.php PLoS] est un journal scientifique couvrant plusieurs disciplines (biologie, physique,...). Les publications ne se font qu'en ligne : il n'y a pas de version papier. Les articles publiés dans PLoS sont indexés dans les bases publiques [http://www.ncbi.nlm.nih.gov/pmc/ PubMed Central], [http://www.scopus.com/home.url Scopus], [http://www.crossref.org/ CrossRef], ainsi que par Google Scholar. | |
| | |
| La philosophie de PLoS est différente de celle des journaux scientifiques classiques tels que Nature, Science,... qui traduisent l'obsession universitaire générale de l'''impact factor'' (réf) du journal. En effet, l'idée est qu'une publication en ligne implique de publier plus rapidement les résultats de sa recherche et de laisser l'évaluation du travail scientifique à une discussion ouverte entre pairs. De plus, le journal ne se limite pas à un domaine scientifique restreint, mais souhaite favoriser et rendre possible la publication d'articles interdisciplinaires. Mais ce qui est encore plus remarquable, c'est la licence des publications : elles sont toutes sous Creative Commons Attribution Licence (CCAL), [http://www.plos.org/journals/license.html la page dédiée à la licence] précisant :
| |
| Under the CCAL, authors retain ownership of the copyright for their article, but authors allow
| |
| anyone to download, reuse, reprint, modify, distribute, and/or copy articles in PLoS journals,
| |
| so long as the original authors and source are cited. No permission is required from the authors
| |
| or the publishers.
| |
| Enfin, vous avez la possibilité de soumettre votre article en tant que fichier LaTeX :-)
| |
| | |
| ===BioMed Central===
| |
| [http://www.biomedcentral.com/ BioMed Central (BMC)] ainsi que Chemistry Central et PhysMath Central sont depuis 2008 propriété de Springer Science ; les articles ne sont publiés qu'en ligne. [http://www.biomedcentral.com/info/about/whatis Leur philosophie] est semblable à celle de PLoS :
| |
| All original research articles published by BioMed Central are made freely and permanently
| |
| accessible online immediately upon publication. BioMed Central views open access to research
| |
| as essential in order to ensure the rapid and efficient communication of research findings.
| |
| Les publications sont indexées dans les bases publiques [http://www.ncbi.nlm.nih.gov/pmc/ PubMed Central], [http://www.scopus.com/home.url Scopus], [http://www.crossref.org/ CrossRef], ainsi que par Google Scholar.
| |
| | |
| BMC publie 204 journaux dont les articles sont sous Creative Commons Attribution Licence (CCAL) et couvrant une large palettes de domaines scientifiques et médicaux. L'accès à certaines revues est ouverte seulement aux universitaires (par exemple, Genome Research).
| |
| | |
| =Quelques articles pour alimenter la réflexion=
| |
| Pour l'instant, juste les titres et les résumés (''abstracts'') sont donnés. La plupart des articles étant en accès payant, les pdf ne sont pas publiés...
| |
| | |
| * '''When speed truly matters, openness is the answer.'''
| |
| | |
| ''Marturano A.''
| |
| | |
| ''Sacred Heart Catholic University of Rome and LUISS University of Rome, Italy. marturanoa A luiss DOT it
| |
| Bioethics. 2009 Sep;23(7):385-93. Epub 2009 Apr 21.''
| |
| | |
| In this paper I analyse the ethical implications of the two main competing methodologies in genomic research. I do not aim to provide another contribution from the mainstream legal and public policy perspective; rather I offer a novel approach in which I analyse and describe the patent-and-publish regime (the proprietary regime) led by biologist J. Craig Venter and the 'open-source' methodologies led by biotechnology Nobel laureate John Sulston. The 'open-source methodologies' arose in biotechnology as an alternative to the patent-and-publish regime in the wake of the explosion in computer technology. Indeed, the tremendous increase in computer technology has generated a corresponding increase in the pace of genomics research. I conclude this paper by arguing that while the patent-and-publish method is a transactional method based on the exchange of extrinsic goods (patents in exchange for research funds), the free and open-source methodology (FLOSS) is a transformational method based on a visionary ideal of science, which leads to prioritizing intrinsic goods in scientific research over extrinsic goods.
| |
| | |
| | |
| * '''Open source and healthcare in Europe - time to put leading edge ideas into practice.'''
| |
| | |
| ''Murray PJ, Wright G, Karopka T, Betts H, Orel A.''
| |
| | |
| ''CHIRAD - Centre for Health Informatics Research and Development, Lincolnshire, UK. peterjmurray A gmail DOT com
| |
| Stud Health Technol Inform. 2009;150:963-7.''
| |
| | |
| Free/Libre and Open Source Software (FLOSS) is a process of software development, a method of licensing and a philosophy. Although FLOSS plays a significant role in several market areas, the impact in the health care arena is still limited. FLOSS is promoted as one of the most effective means for overcoming fragmentation in the health care sector and providing a basis for more efficient, timely and cost effective health care provision. The 2008 European Federation for Medical Informatics (EFMI) Special Topic Conference (STC) explored a range of current and future issues related to FLOSS in healthcare (FLOSS-HC). In particular, there was a focus on health records, ubiquitous computing, knowledge sharing, and current and future applications. Discussions resulted in a list of main barriers and challenges for use of FLOSS-HC. Based on the outputs of this event, the 2004 Open Steps events and subsequent workshops at OSEHC2009 and Med-e-Tel 2009, a four-step strategy has been proposed for FLOSS-HC: 1) a FLOSS-HC inventory; 2) a FLOSS-HC collaboration platform, use case database and knowledge base; 3) a worldwide FLOSS-HC network; and 4) FLOSS-HC dissemination activities. The workshop will further refine this strategy and elaborate avenues for FLOSS-HC from scientific, business and end-user perspectives. To gain acceptance by different stakeholders in the health care industry, different activities have to be conducted in collaboration. The workshop will focus on the scientific challenges in developing methodologies and criteria to support FLOSS-HC in becoming a viable alternative to commercial and proprietary software development and deployment.
| |
| | |
| | |
| * '''osni.info-Using free/libre/open source software to build a virtual international community for open source nursing informatics.'''
| |
| | |
| ''Oyri K, Murray PJ.''
| |
| | |
| ''The Interventional Centre, Rikshospitalet University Hospital, 0027 Oslo, Norway. karl.oyri A klinmed DOT uio DOT no''
| |
| ''Int J Med Inform. 2005 Dec;74(11-12):937-45. Epub 2005 Aug 19.''
| |
| | |
| Many health informatics organizations seem to be slow to take up the advantages of dynamic, web-based technologies for providing services to, and interaction with, their members; these are often the very technologies they promote for use within healthcare environments. This paper aims to introduce some of the many free/libre/open source (FLOSS) applications that are now available to develop interactive websites and dynamic online communities as part of the structure of health informatics organizations, and to show how the Open Source Nursing Informatics Working Group (OSNI) of the special interest group in nursing informatics of the International Medical Informatics Association (IMIA-NI) is using some of these tools to develop an online community of nurse informaticians through their website, at . Some background introduction to FLOSS applications is used for the benefit of those less familiar with such tools, and examples of some of the FLOSS content management systems (CMS) being used by OSNI are described. The experiences of the OSNI will facilitate a knowledgeable nursing contribution to the wider discussions on the applications of FLOSS within health and healthcare, and provides a model that many other groups could adopt.
| |
| | |
| | |
| * '''Open Source software in medical informatics--why, how and what.'''
| |
| | |
| ''McDonald CJ, Schadow G, Barnes M, Dexter P, Overhage JM, Mamlin B, McCoy JM.''
| |
| | |
| ''Department of Medicine, Indiana University School of Medicine, 1001 W. 10th St., RG5th Floor, Indianapolis, IN 46202, USA. cmcdonald A regenstrief DOT org. Int J Med Inform. 2003 Mar;69(2-3):175-84. Comment in: Int J Med Inform. 2003 Sep;71(2-3):165.''
| |
| | |
| 'Open Source' is a 20-40 year old approach to licensing and distributing software that has recently burst into public view. Against conventional wisdom this approach has been wildly successful in the general software market--probably because the openness lets programmers the world over obtain, critique, use, and build upon the source code without licensing fees. Linux, a UNIX-like operating system, is the best known success. But computer scientists at the University of California, Berkeley began the tradition of software sharing in the mid 1970s with BSD UNIX and distributed the major internet network protocols as source code without a fee. Medical informatics has its own history of Open Source distribution: Massachusetts General's COSTAR and the Veterans Administration's VISTA software have been distributed as source code at no cost for decades. Bioinformatics, our sister field, has embraced the Open Source movement and developed rich libraries of open-source software. Open Source has now gained a tiny foothold in health care (OSCAR GEHR, OpenEMed). Medical informatics researchers and funding agencies should support and nurture this movement. In a world where open-source modules were integrated into operational health care systems, informatics researchers would have real world niches into which they could engraft and test their software inventions. This could produce a burst of innovation that would help solve the many problems of the health care system. We at the Regenstrief Institute are doing our part by moving all of our development to the open-source model.
| |
| | |
| | |
| * '''How the role of computing is driving new genetics' public policy.'''
| |
| | |
| ''Marturano A, Chadwick R.''
| |
| | |
| ''Centre for Leadership Studies, School of Business and Economics, University of Exeter, Streatham Court, Reenes Drive, Exeter EX 4 4PU, UK. antonio.marturano A ex DOT ac DOT uk. Ethics Inf Technol. 2004;6(1):43-53.''
| |
| | |
| In this paper we will examine some ethical aspects of the role that computers and computing increasingly play in new genetics. Our claim is that there is no new genetics without computer science. Computer science is important for the new genetics on two levels: (1) from a theoretical perspective, and (2) from the point of view of geneticists practice. With respect to (1), the new genetics is fully impregnate with concepts that are basic for computer science. Regarding (2), recent developments in the Human Genome Project (HGP) have shown that computers shape the practices of molecular genetics; an important example is the Shotgun Method's contribution to accelerating the mapping of the human genome. A new challenge to the HGP is provided by the Open Source Philosophy (I computer science), which is another way computer technologies now influence the shaping of public policy debates involving genomics.
| |
| | |
| | |
| | |
| * '''The case for open-source software in drug discovery.'''
| |
| | |
| ''DeLano WL.''
| |
| | |
| ''DeLano Scientific LLC, 400 Oyster Point Blvd., Suite 213, San Francisco, CA 94080, USA. Drug Discov Today. 2005 Feb 1;10(3):213-7.''
| |
| | |
| Widespread adoption of open-source software for network infrastructure, web servers, code development, and operating systems leads one to ask how far it can go. Will "open source" spread broadly, or will it be restricted to niches frequented by hopeful hobbyists and midnight hackers? Here we identify reasons for the success of open-source software and predict how consumers in drug discovery will benefit from new open-source products that address their needs with increased flexibility and in ways complementary to proprietary options.
| |
| | |
| | |
| * '''The role of open-source software in innovation and standardization in radiology.'''
| |
| | |
| ''Erickson BJ, Langer S, Nagy P.''
| |
| | |
| ''Department of Radiology, Radiology Informatics Laboratory, Mayo Clinic, Rochester, MN 55905, USA. bje A mayo DOT edu''
| |
| | |
| The use of open-source software (OSS), in which developers release the source code to applications they have developed, is popular in the software industry. This is done to allow others to modify and improve software (which may or may not be shared back to the community) and to allow others to learn from the software. Radiology was an early participant in this model, supporting OSS that implemented the ACR-National Electrical Manufacturers Association (now Digital Imaging and Communications in Medicine) standard for medical image communications. In radiology and in other fields, OSS has promoted innovation and the adoption of standards. Popular OSS is of high quality because access to source code allows many people to identify and resolve errors. Open-source software is analogous to the peer-review scientific process: one must be able to see and reproduce results to understand and promote what is shared. The authors emphasize that support for OSS need not threaten vendors; most vendors embrace and benefit from standards. Open-source development does not replace vendors but more clearly defines their roles, typically focusing on areas in which proprietary differentiators benefit customers and on professional services such as implementation planning and service. Continued support for OSS is essential for the success of our field.
| |
| | |
| | |
| * '''Public vs. proprietary science: a fruitful tension?'''
| |
| | |
| ''Eisenberg RS, Nelson RR.''
| |
| | |
| ''University of Michigan Law School, Ann Arbor, Michigan, USA. Acad Med. 2002 Dec;77(12 Pt 2):1392-9.''
| |
| | |
| The authors examine the presumption that basic scientific research is most effectively utilized when the findings of that research are openly disseminated without significant restriction, while research with more practical application should be the prerogative of private enterprise. However, many fields, including molecular biology generally and genomics in particular, lie in the intersection between basic research and application. Moreover, institutional boundaries that once reasonably sharply demarcated basic research from technological development have grown porous, with more academic research finding application in industry. The authors consider the Human Genome Project and rival industry sequencing efforts as a case in point of the new political economy of scientific research. Since the inception of the Human Genome Project, there has been general agreement among researchers that the project would be most advantageous to science if the sequence data were made publicly available, quickly and without restriction. Many of these arrangements required federal agencies and some universities to "maneuver around" the Bayh-Dole Act. In several cases, most notably genomic sequences and the SNPs (i.e., single nucleotide polymorphisms) consortium, it was the pharmaceutical industry that initiated or helped enable the project to ensure open and unencumbered access to information, the type of access that has historically been the provenance of academia and the raison d'être of academic research. The authors conclude by reasserting the value of public science as a broadly valuable and enabling social commitment, not limited simply to the products or technologies it spawns.
| |
| | |
| | |
| * '''The state and profile of open source software projects in health and medical informatics.'''
| |
| | |
| ''Janamanchi B, Katsamakas E, Raghupathi W, Gao W.''
| |
| | |
| ''Texas A&M International University, Laredo, Texas, USA. Balaji.janamanchi A gmail DOT com. Int J Med Inform. 2009 Jul;78(7):457-72. Epub 2009 Mar 25.''
| |
| | |
| PURPOSE: Little has been published about the application profiles and development patterns of open source software (OSS) in health and medical informatics. This study explores these issues with an analysis of health and medical informatics related OSS projects on SourceForge, a large repository of open source projects. METHODOLOGY: A search was conducted on the SourceForge website during the period from May 1 to 15, 2007, to identify health and medical informatics OSS projects. This search resulted in a sample of 174 projects. A Java-based parser was written to extract data for several of the key variables of each project. Several visually descriptive statistics were generated to analyze the profiles of the OSS projects. RESULTS: Many of the projects have sponsors, implying a growing interest in OSS among organizations. Sponsorship, we discovered, has a significant impact on project success metrics. Nearly two-thirds of the projects have a restrictive license type. Restrictive licensing may indicate tighter control over the development process. Our sample includes a wide range of projects that are at various stages of development (status). Projects targeted towards the advanced end user are primarily focused on bio-informatics, data formats, database and medical science applications. CONCLUSION: We conclude that there exists an active and thriving OSS development community that is focusing on health and medical informatics. A wide range of OSS applications are in development, from bio-informatics to hospital information systems. A profile of OSS in health and medical informatics emerges that is distinct and unique to the health care field. Future research can focus on OSS acceptance and diffusion and impact on cost, efficiency and quality of health care.
| |
| | |
| | |
| * '''Developing Online Communities with LAMP (Linux, Apache, MySQL, PHP) - the IMIA OSNI and CHIRAD Experiences.'''
| |
| | |
| ''Murray PJ, Oyri K.''
| |
| | |
| ''Centre for Health Informatics Research and Development (CHIRAD), UK. Stud Health Technol Inform. 2005;116:361-6.''
| |
| | |
| Many health informatics organisations do not seem to use, on a practical basis, for the benefit of their activities and interaction with their members, the very technologies that they often promote for use within healthcare environments. In particular, many organisations seem to be slow to take up the benefits of interactive web technologies. This paper presents an introduction to some of the many free/libre and open source (FLOSS) applications currently available and using the LAMP - Linux, Apache, MySQL, PHP architecture - as a way of cheaply deploying reliable, scalable, and secure web applications. The experience of moving to applications using LAMP architecture, in particular that of the Open Source Nursing Informatics (OSNI) Working Group of the Special Interest Group in Nursing Informatics of the International Medical Informatics Association (IMIA-NI), in using PostNuke, a FLOSS Content Management System (CMS) illustrates many of the benefits of such applications. The experiences of the authors in installing and maintaining a large number of websites using FLOSS CMS to develop dynamic, interactive websites that facilitate real engagement with the members of IMIA-NI OSNI, the IMIA Open Source Working Group, and the Centre for Health Informatics Research and Development (CHIRAD), as well as other organisations, is used as the basis for discussing the potential benefits that could be realised by others within the health informatics community.
| |
|
| |
|
| | =Journaux libres pour la science= |
| | Voir la page [[Publications scientifiques]] |
|
| |
|
| = Liens = | | = Liens = |
| Ligne 227 : |
Ligne 143 : |
| * Un groupe de travail pour la promotion et le développement des logiciels libres en Sciences : http://fsffrance.org/science/science.fr.html | | * Un groupe de travail pour la promotion et le développement des logiciels libres en Sciences : http://fsffrance.org/science/science.fr.html |
| * Un projet pour la promotion et le développement des logiciels libres en Chimie : http://www.alchem.org | | * Un projet pour la promotion et le développement des logiciels libres en Chimie : http://www.alchem.org |
| * Un projet de diffusion libre de litérature grise (thèses, rapports, etc.) : http://bibliotheques.univ-lille1.fr/grisemine | | * Un projet de diffusion libre de littérature grise (thèses, rapports, etc.) : http://bibliotheques.univ-lille1.fr/grisemine |
| * Un projet pour faciliter l'écriture et la publication avec des Logiciels Libres de rapports, thèses, etc. : http://sourcesup.cru.fr/cybertheses/ | | * Un projet pour faciliter l'écriture et la publication avec des Logiciels Libres de rapports, thèses, etc. : http://sourcesup.cru.fr/cybertheses/ |
| * Voir aussi GNU eprints : http://www.eprints.org/ | | * Voir aussi GNU eprints : http://www.eprints.org/ |
| * Des vidéos scientifiques disponibles gratuitement (mais dans un format fermé Flash) : http://www.canal-u.tv/ | | * Des vidéos scientifiques disponibles gratuitement (mais dans un format fermé Flash) : http://www.canal-u.tv/ |
| | * Fiches descriptives de logiciels, la plupart libres, utilisés ou développés dans l' Enseignement Supérieur et la Recherche : http://www.projet-plume.org/ |
|
| |
|
| [[Catégorie:Science]] | | [[Catégorie:Science]] |